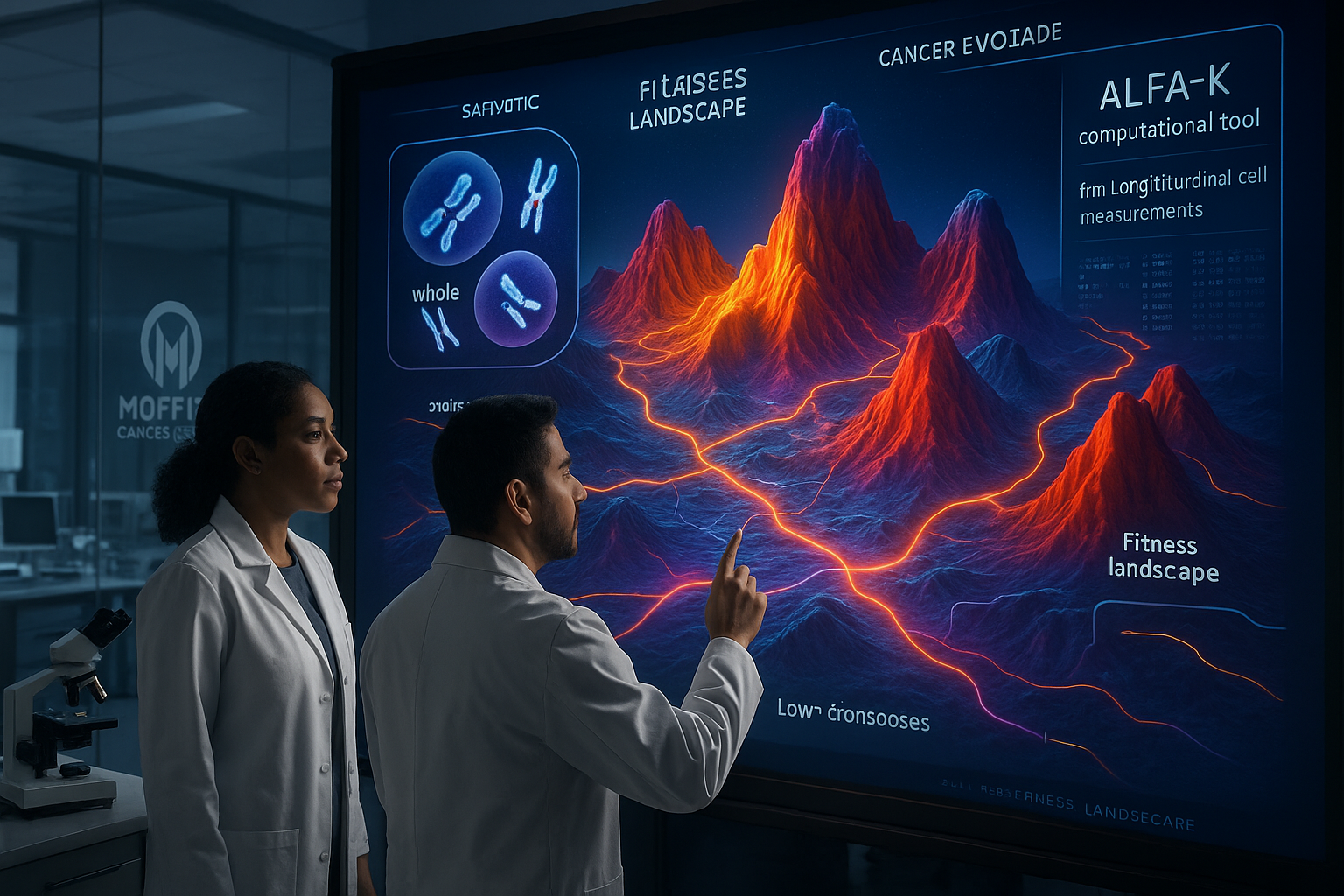
Des scientifiques du Moffitt Cancer Center rapportent le développement d’une méthode computationnelle, ALFA-K, qui utilise des mesures longitudinales de cellules uniques pour inférer comment les gains et pertes de chromosomes entiers peuvent façonner le chemin évolutif d’une tumeur. Ce travail, publié dans Nature Communications, soutient que ces changements chromosomiques à grande échelle suivent des schémas mesurables influencés par le contexte cellulaire et le stress lié au traitement plutôt que de se dérouler comme une pure aléatoire.
Le cancer peut évoluer rapidement lorsque les cellules gagnent ou perdent des chromosomes entiers — des changements à grande échelle qui modifient la dosage de nombreux gènes à la fois et peuvent remodeler la façon dont une tumeur croît et répond au stress. Des chercheurs du H. Lee Moffitt Cancer Center & Research Institute décrivent une nouvelle approche computationnelle, appelée ALFA-K (Adaptive Local Fitness landscapes for Aneuploid Karyotypes), visant à prédire comment de tels changements au niveau chromosomique s’accumulent au fil du temps. L’équipe affirme que ALFA-K est conçu pour les données longitudinales de cellules uniques, lui permettant de reconstruire comment les populations de cellules cancéreuses passent par différents « états » chromosomiques et d’estimer quelles combinaisons de chromosomes semblent favorisées sous des pressions de sélection, y compris le stress lié au traitement. Dans un Q&R accompagnant la sortie, l’auteure principale Noemi Andor a déclaré que l’objectif était de dépasser les instantanés uniques de la génétique tumorale et de quantifier quelles combinaisons de chromosomes aident les cellules à persister. « Le cancer évolue. Alors que les tumeurs grandissent, leurs cellules commettent constamment des erreurs lors de la copie et de la division de leur ADN. Beaucoup de ces erreurs impliquent de gagner ou de perdre des chromosomes entiers », a dit Andor. Selon les chercheurs, ALFA-K diffère des approches antérieures qui traitaient souvent les gains ou pertes individuels de chromosomes comme ayant des effets fixes. Au lieu de cela, il modélise la dépendance au contexte — le même changement chromosomique peut être avantageux ou nuisible selon la composition chromosomique existante de la cellule — et intègre l’instabilité chromosomique continue. Dans l’étude, l’équipe rapporte l’estimation de la fitness à travers plus de 270 000 configurations chromosomiques distinctes, et conclut que les conditions environnementales et le traitement au cisplatine peuvent changer l’impact fitness des changements de nombre de copies chromosomiques. L’analyse met également en lumière le doublement du génome entier — quand une cellule duplique tous ses chromosomes — comme un mécanisme qui peut atténuer certains des méfaits de l’instabilité chromosomique extrême, les chercheurs décrivant un seuil au-delà duquel le doublement génomique devient avantageux sur le plan évolutif. L’article liste Richard J. Beck, Tao Li et Noemi Andor comme auteurs et a été publié en ligne dans Nature Communications fin décembre 2025. Le travail a été soutenu par le U.S. National Cancer Institute, y compris les subventions 1R37CA266727-01A1, 1R21CA269415-01A1 et 1R03CA259873-01A1. Les chercheurs disent que, à long terme, des outils comme ALFA-K pourraient soutenir des stratégies de traitement « conscientes de l’évolution » — en utilisant des échantillonnages répétés comme des biopsies pour identifier quand les tumeurs pourraient approcher des transitions évolutives risquées et sélectionner des thérapies destinées à limiter les voies vers la résistance aux médicaments.